Background.
The sequencing of the human genome has revealed that almost half of the genome consists of interspersed repeats. Among the different types of interspersed repeats in humans, Alu elements are the most frequent ones, and new Alu elements are still integrated into new positions in the humane genome. In this review some consequences of this Alu activity are discussed. The first part of the review is focused on Alu element activity that causes genetic disease while the use of Alu elements as genetic tools for several purposes is discussed in the last part.
Materials and methods.
This review article is based on a literature search on Medline.
Results and interpretation.
Alu elements cause disease by two different mechanisms. A de novo insertion of an Alu element into, or close to, a gene may disrupt its normal function, while homologous recombination between two Alu elements may result in deletions or duplications that cause genetic disease.
The high density of Alu elements in the human genome combined with their absence in non-primate species make them useful as genetic tools. Alu elements also represent «molecular fossils» that give phylogenetic information about primates as well as information about historical DNA rearrangements in the human genome.
Alu-elementer tilhører en gruppe DNA-sekvenser som kalles innskutt repetitivt DNA. De har økt i antall over tid ved å kopiere seg selv. Disse elementene er også blitt kalt «egoistisk DNA», siden de tilsynelatende mangler en generell funksjon. Mer enn 45 % av vårt genom består av innskutt repetitivt DNA – det er et lappeteppe av relativt korte unike DNA-sekvenser, hyppig avbrutt av ulike typer innskutt repetitivt DNA (1, 2). Tabell 1 viser de ulike hovedgruppene av innskutt repetitivt DNA i det humane genom. Alu-elementer klassifiseres sammen med andre korte repetitive elementer i en gruppe som kalles SINE (short interspersed elements) (2). I denne oversiktsartikkelen belyses spesielt den genteknologiske nytten av Alu-elementer samt hvordan deres aktivitet kan føre til genetisk sykdom. Oversikten som presenteres er basert på en litteraturstudie av orginalartikler fra Medline.
Tabell 1
Innskutt repetitivt DNA i det humane genom
| Hovedgruppe |
Antall¹ |
Andel² (%) |
Undergrupper |
| Korte repetitive elementer (SINE) |
1,6 · 10⁶ |
13 |
Alu, MIR, MIR3 |
| Lange repetitive elementer (LINE) |
0,9 · 10⁶ |
21 |
LINE 1, 2, 3 |
| Retrovirusliknende |
0,5 · 10⁶ |
8 |
ERV I-III, MaLR |
| DNA-transposoner |
0,3 · 10⁶ |
3 |
MER1, MER2 |
| Uklassifiserte |
0,03 · 10⁶ |
0,15 |
Uklassifiserte |
| [i] | |||
[i] ¹ Antall kopier i det (haploide) humane genom
² Andel i prosent av det (haploide) humane genom
Alu-elementene (fig 1) ble identifisert for mer enn 20 år siden. De består av en 300 basepar lang sekvens som kuttes i to av restriksjonsenzymet Alu-I, derav navnet Alu-elementer (1). Elementene har økt i antall ved en prosess som kalles retroposisjon. Denne starter ved at en RNA-polymerase III transkriberer Alu-elementet slik at det dannes et Alu-RNA. Alu-elementets egne RNA-polymerase III-promotorer (fig 1, boks A og boks B) er med på å initiere transkripsjonen (prosess der en RNA-kopi dannes fra DNA), og promotorene blir selv en del av RNA-transkriptet. Ved hjelp av enzymene reverstranskriptase og endonuklease vil det dannes et nytt Alu-element (DNA) fra Alu-RNA-transkriptet. Det integreres i en ny posisjon i genomet uavhengig av posisjonen til det orginale Alu-elementet. Den siste delen av retroposisjon er lite karakterisert, men antas å være avhengig av poly-A-halen på Alu-elementet (fig 1), mens enzymene «lånes» fra LINE-gruppen. Alu-elementene er med andre ord avhengig av en annen gruppe innskutt repetitivt DNA (tab 1, LINE, long interspersed elements) for retroposisjon, de blir derfor også omtalt som parasittens parasitter (1).

Millioner av år med retroposisjon har ført til en betydelig akkumulering av Alu-elementer. I det (haploide) humane genom er det i snitt ett slikt element for hver tredje kilobase DNA (ca. 1,1 millioner kopier). De er dermed den hyppigst forekommende type innskutt repetitivt DNA i mennesker (2). Et karakteristisk trekk ved dem er at de finnes i alle primater, men ikke i andre arter. Man antar at en fusjon mellom to RNA-gener for 65 – 80 millioner år siden var opprinnelsen til Alu-elementene (1).
Noen få Alu-elementer er fremdeles aktive. Basert på grove beregninger er det anslått at det er 1 – 10 nye Alu-insersjoner (et Alu-element som settes inn i en ny posisjon i genomet) per 200 fødsler (3). Enkelte Alu-elementer har adoptert viktige funksjoner. Noen deltar i reguleringen av gentranskripsjon, andre i spleising av pre-mRNA (en klippe-og-lime-modifisering av pre-mRNA). I enkelte tilfeller har Alu-elementer også gitt gener nye funksjoner (4).
Alu-elementer og sykdom
Sykdom forårsaket av insersjoner
Det er et faktum at Alu-elementene fremdeles øker i antall og integreres i nye posisjoner. Den mest nærliggende sykdomsmekanismen er derfor insersjon av et Alu-element i eller nær et gen. En insersjon i et ekson vil ødelegge det affiserte genets normale funksjon siden Alu-elementet både forandrer genets leseramme og legger til en sekvens som vil gi 100 ekstra aminosyrer i proteinet.
Et klassisk eksempel på en slik Alu-insersjon er et tilfelle av alvorlig hemofili A hos to brødre i en familie uten tidligere forekomst av hemofili (tab 2, nr. 1) (5). Hemofili A skyldes defekt eller mangel på faktor VIII. Sukarova og medarbeidere viste ved å sekvensere faktor VIII-genet at de to brødrene hadde en Alu-insersjon i ekson 14, og at deres mor og mormor var bærere av denne mutasjonen. Den innskutte Alu-sekvensen inneholdt et stoppkodon som resulterte i for tidlig terminering av translasjonen (en prosess der en polypeptidkjede dannes ved hjelp av informasjon fra mRNA) og dermed et trunkert funksjonsløst protein.
Tabell 2
Alu-insersjoner som har forårsaket eller bidratt til ulike genetiske sykdommer
| Sykdom¹ |
Forekomst |
Gen |
PubMed-indeksnummer² |
|
| 1. |
Hemofili A |
De novo? |
Factor VIII |
11713379 |
| 2. |
Hemofili B |
Familiær, de novo |
Faktor IX |
8069649, 11385709, 10679958 |
| 3. |
Arvelig desmoid tumor |
Familiær |
APC |
10077730 |
| 4. |
Nevrofibromatose type 1 |
De novo |
NF1 |
1719426 |
| 5. |
Ornitinaminotransferasedefekt |
Ukjent |
OAT |
1992472 |
| 6. |
Alports syndrom |
Familiær |
COL4A3 |
7633417 |
| 7. |
Aperts syndrom |
De novo |
FGFR2 |
9973282 |
| 8. |
Akolinesterasemi |
Familiær |
Kolinesterase |
1662391 |
| 9. |
Komplementsvikt |
De novo |
C1-inhibitor |
2154751 |
| 10. |
Hyperparatyreoisme, hyperkalsemi |
Familiær |
CaR |
7717399 |
| 11. |
Brankio-oto-renalt syndrom |
De novo |
EYA1 |
9361030 |
| 12. |
Brystkreft |
De novo |
BRCA2 |
8640237 |
| 13. |
Akutt porfyri |
Familiær |
PBGD |
10408772 |
| 14. |
Glyserolkinasedefekt |
Ukjent |
GK |
10737976 |
| 15. |
Autoimmunt lymfoproliferativt syndrom |
Familiær |
Fas (apo-1) |
12215906 |
| [i] | ||||
[i] ¹ Tabellen inneholder ulike eksempler og representerer ikke en fullstendig liste over sykdommer forårsaket av Alu-insersjon
² PubMed-indeksnummer gir direkte tilgang på referanser og sammendrag av orginalartiklene i tabellen ved søk i PubMed (5)
Nærmere undersøkelser av årsaken til en rekke ulike genetiske sykdommer, f.eks. hemofili B og arvelig desmoid tumor (familiær adenomatøs polypose), har vist at sykdommene er forårsaket av liknende Alu-insersjoner i eksoner (tab 2, nr. 2, 3).
Insersjoner av Alu-elementer i introner nær eksoner kan også forårsake sykdom ved å forandre spleisingen av pre-mRNA slik at et ekson utelates fra det ferdig prosesserte mRNA (exon skipping) (tab 2, nr. 4).
Deler av Alu-elementet, såkalte kryptiske spleiseseter, har stor likhet med sekvenser som regulerer spleising. Et nøytralt Alu-element i et gitt intron kan dermed over tid forandres til en aktiv regulator av spleising ved at en enkelt baseparmutasjon forandrer det kryptiske spleisesetet til en aktiv spleiseregulator som forårsaker genetisk sykdom (tab 2, nr. 5, 6).
Tabell 2 viser genetiske sykdommer forårsaket av eller mest sannsynlig forårsaket av Alu-insersjoner. Grove estimater basert på rapportering av slike mutasjoner til databaser antyder at rundt 0,1 % av genetiske sykdommer skyldes Alu-insersjoner (3).
Sykdom forårsaket av rekombinasjon
Homolog rekombinasjon sørger for korrekt fordeling av kromosomer i meiosen og for resiprok (gjensidig) utveksling av arvemateriale mellom to homologe kromosomer. Homolog rekombinasjon starter med en baseparing av identiske sekvenser fra komplementære enkelttråder i de to homologe kromosomer. Uten en vellykket baseparing mellom de komplementære enkelttrådene avbrytes rekombinasjonen. Baseparingen sørger for at homolog rekombinasjon vanligvis initieres mellom alleliske sekvenser i de to homologe kromosomene. Rekombinasjon er derfor en meget nøyaktig prosess der det verken legges til eller trekkes fra et eneste basepar (6).
To tilfeldige Alu-elementer har en gjennomsnittlig sekvenslikhet på 85 – 90 % (7), og det er omtrent ett Alu-element for hvert 3 Kb DNA. Det er derfor tallrike muligheter for at generell rekombinasjon kan initieres mellom to ikke-alleliske Alu-elementer siden de har nærmest identiske sekvenser. En slik ikke-allelisk homolog rekombinasjon vil imidlertid ikke være resiprok. Isteden vil den resultere i ett kromosom med en duplisering av området mellom Alu-elementene og ett kromosom med en delesjon (tap) av det samme området (fig 2).
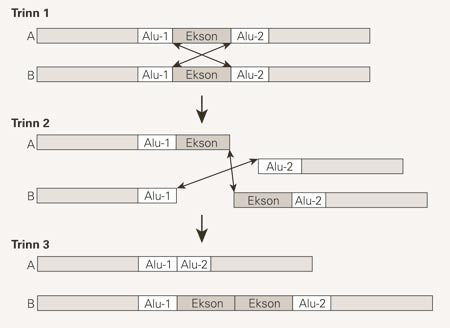
Grove estimater basert på rapporteringer til mutasjonsdatabaser viser at rundt 0,3 % av genetiske sykdommer skyldes slike Alu-rekombinasjoner (3). Dermed synes det som om denne mekanismen er en hyppigere årsak til sykdom enn Alu-insersjoner. Tabell 3 viser eksempler på genetiske sykdommer der Alu-rekombinasjon enten er den direkte årsaken til eller har bidratt til sykdommen.
Tabell 3
Oversikt over Alu-rekombinasjoner som har forårsaket eller bidratt til ulike genetiske sykdommer
| Sykdom¹ |
Mekanisme |
Gen |
PubMed-indeksnummer² |
|
| 1. |
Hyperkolesterolemi |
Intragenisk Alu-rekombinasjon |
LDLR |
3155573, 3815525, 2544509 |
| 2. |
Angionevrotisk ødem |
Intragenisk Alu-rekombinasjon |
C1-inhibitor |
2572212, 2276734 |
| 3. |
Tay-Sachs’ sykdom |
Intragenisk Alu-rekombinasjon |
b-HEXA |
2824459 |
| 4. |
Insulinuavhengig diabetes |
Intragenisk Alu-rekombinasjon |
Ins. Rec. B |
1971035 |
| 5. |
Arvelig C3-defekt |
Intragenisk Alu-rekombinasjon |
C3 |
1350678 |
| 6. |
Sandhoffs sykdom |
Intragenisk Alu-rekombinasjon |
HEXB |
2147027 |
| 7. |
Hypobetalipoproteinemi |
Intragenisk Alu-rekombinasjon |
apoB |
2567736 |
| 8. |
Alvorlig kombinert immunsvikt (SCID) |
Intragenisk Alu-rekombinasjon |
ADA |
3366897, 1696926 |
| 9. |
Arvelig tykktarmskreft (HNPCC) |
Intragenisk Alu-rekombinasjon |
hMSH2 |
12494471 |
| 10. |
Arvelig tykktarmskreft (HNPCC) |
Intragenisk Alu-rekombinasjon |
hMLH1 |
7584997, 8971183 |
| 11. |
B-cellelymfom |
Intragenisk Alu-rekombinasjon |
p107 (Rb) |
10863094 |
| 12. |
Hunters syndrom |
Intragenisk Alu-rekombinasjon |
IDS |
12579417 |
| 13. |
Fabrys sykdom |
Intragenisk Alu-rekombinasjon |
α-gal A |
2160973 |
| 14. |
α-talassemi |
Subtelomere Alu-rearrangement |
α-globin |
3032452, 8842736, 9462544 |
| 15. |
Glanzmanns trombobasteni |
Alu-inversjon/-delesjon |
Integrin |
8317479 |
| 16. |
Ehlers-Danlos syndrom |
Alu-rekombinasjon. Stor duplikasjon |
Lysinhydroksylase |
7977351 |
| 17. |
Duchennes muskeldystrofi |
Alu-rekombinasjon. Meget stor duplikasjon |
DMD |
1868831 |
| 18. |
Lesch-Nyhans syndrom |
Alu-rekombinasjon. Stor duplikasjon |
HPRT |
8381385 |
| 18. |
Li-Fraumenis syndrom |
Alu-rekombinasjon. Stor delesjon |
TP53 |
12584563 |
| 19. |
XX-mann |
Alu-XY-rekombinasjon |
XY |
2822256 |
| 20. |
Brystkreft |
Intragenisk Alu-rekombinasjon samt somatisk |
BRCA1 |
9041180, 9285788 |
| 21. |
Akutt myeloid leukemi |
Translokasjon t(8;16) |
MOZ/CBP |
12461753 |
| 22. |
Pelizaeus-Merzbackers sykdom |
Translokasjon t(19;X) |
PLP1 |
12297985 |
| 23. |
Akutt lymfoblastisk leukemi |
Somatiske alu-re-arrangem. |
ALL-1 |
8044771, 9482895, 8988051 |
| 24. |
Ewings sarkom |
Somatisk interkromosomal alu-rek |
TRE |
1461655 |
| [i] | ||||
[i] ¹ Tabellen inneholder ulike eksempler, og representerer ikke en fullstendig liste over sykdommer forårsaket av Alu-rekombinasjon
² PubMed-indeksnummer gir direkte tilgang på referanser og sammendrag av orginalartiklene i tabellen ved søk i PubMed (5)
Mindre duplikasjoner eller delesjoner forårsaket av rekombinasjon mellom ikke-alleliske Alu-elementer innen et gitt gen (intrageniske mutasjoner) er den hyppigste typen Alu-rekombinasjon (tab 3, nr. 1 – 13), men dersom det er stor avstand mellom to Alu-elementer som rekombinerer, kan dette føre til større rearrangementer, som duplikasjon av et fragment på over 100 Kb (tab 3, nr. 17).
Alu-rekombinasjon mellom elementer på ulike kromosomer kan føre til translokasjoner som gir genetisk sykdom. Eksempler på dette er en translokasjon som har resultert i en fusjon mellom genene MOZ (histonacetyltransferase) og CBP (CREB-bindende protein) samt en translokasjon som har ført til tap av genet PLP1 (tab 3, nr. 21, 22).
I noen gener er det rapportert mange uavhengige Alu-rekombinasjoner (tab 3, nr. 1, 2). Hva som er årsaken til en høyere rate av Alu-rekombinasjon i enkelte gener er ukjent, og det er ikke funnet noen direkte sammenheng mellom høy tetthet av Alu-elementer i et gitt gen og høy rate av Alu-rekombinasjon (P. Deininger, personlig meddelelse). Et annet gen med multiple rearrangementer forårsaket av Alu-elementer er ALL-1 (akutt lymfoblastisk leukemi) (tab 3, nr. 23). Mutasjonene i ALL-1 skiller seg imidlertid fra de tidligere nevnte Alu-rekombinasjonene ved at rearrangeringen foregår i somatiske celler.
Alu-elementer og kreft
Både Alu-insersjoner og Alu-rekombinasjoner kan bidra til utvikling av kreft ved å inaktivere tumorsuppressorgener eller «mismatch»-reparasjonsgener (feilparingsreparasjonsgener). I tumorsuppressorgenet BRCA1 (tab 3, nr. 20) er det vist at en Alu-rekombinasjon som resulterte i en delesjon av ekson 17 førte til inaktivering av genet, mens en Alu-insersjon som førte til alternativ spleising og «exon skipping» har forårsaket inaktivering av tumorsupressorgenet BRCA2 (tab 2, nr. 12). Inaktivering av de nevnte gener (BRCA1 og BRCA2) bidrar til utvikling av brystkreft.
Inaktiverende delesjoner som følge av Alu-rekombinasjon er også funnet i «mismatch»-reparasjonsgenene hMLH1 og hMSH2 (tab 3, nr. 9, 10). Blant annet synes en inaktivering av hMLH1 å være spesielt utbredt blant finner. I en studie av Nystrom-Lahti og medarbeidere ble denne mutasjonen funnet i ca. 40 % av alle tilfeller med arvelig predisposisjon for tykktarmskreft i Finland (8).
Alu-rekombinasjon kan også være en bidragsyter til tap av heterozygositet (LOH), en generell økning av delesjoner som observeres i kreftceller. Mekanismen for dette er knyttet til P53. Ved inaktivering av P53 er det vist at Alu-rekombinasjonsraten øker 20 ganger (9). En slik øking av rekombinasjonsraten fører til en tilsvarende økning av antall delesjoner. Det er derfor sannsynlig at Alu-rekombinasjon bidrar sterkt til tap av heterozygositet i kreftceller.
De fleste eksemplene på sykdomsfremkallende Alu-elementer i tabell 2 og tabell 3 beskriver de novo-mutasjoner, andre er mutasjoner som representerer familiære genetiske sykdommer eller historiske mutasjoner som i dag er relativt hyppige i enkelte populasjoner (f.eks. mutasjonen i hMLH1 i finner). Man kan også tenke seg at det finnes prehistoriske gendefekter som er forårsaket av Alu-elementer for så lenge siden at de er fiksert i hele den humane populasjon. Et fascinerende eksempel på dette synes å være tap av evnen til å syntetisere vitamin C. Genet GLO (gulonolaktonoksidase) koder for et enzym som katalyserer det siste trinnet i syntesen av vitamin C. En studie av Challem & Taylor indikerer at Alu-elementaktivitet kan være årsaken til tapet av dette genet i høyere primater for rundt 45 millioner år siden (10).
Alu-elementer som molekylære verktøy
Tallrike og primatspesifikke elementer
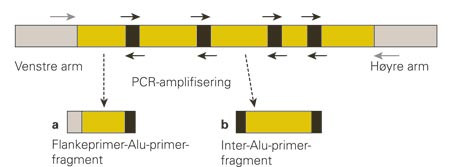
Alu-sekvensen har en kompleksitet som er tilstrekkelig til at både prober og primere (polymerasekjedereaksjonsmetoder, PCR) binder spesifikt til Alu-elementer. Det enorme antallet Alu-elementer kombinert med at de bare er til stede i primatgenomer kan derfor utnyttes i en rekke applikasjoner der man ønsker å detektere eller ekstrahere den humane komponenten i en blandingsprøve. Alu-elementenes spesielle egenskaper kan bl.a. utnyttes ved PCR-amplifisering av den klonede humane komponenten i Yeast Artificial Clones (YAC) (11). Prinsippet for denne metoden er illustrert i figur 3. Ved å benytte PCR-primere som binder spesifikt til Alu-elementer som er til stede i det klonede humane DNA kan man amplifisere den humane komponenten fra en en blanding av humant DNA og gjær-DNA. Ved hjelp av liknende metoder ble de første humane onkogener isolert (12).
De primatspesifikke egenskapene til Alu-elementer kan også utnyttes i rettsgenetikk. Det biologiske prøvematerialet er ofte en blanding av humant DNA og DNA fra andre arter (bakterier, hund, katt). Ved å måle andelen Alu-elementer i prøven kan andelen humant DNA beregnes (13).
Alu-elementer er molekylære fossiler
Retroposisjon av Alu-elementer startet tidlig i primatenes utvikling. Et element som er blitt integrert i en bestemt posisjon, vil arves av alle etterkommere av dette individet, siden det ikke er noen mekanisme som presist fjerner et Alu-element. Det er lite sannsynlig at to Alu-elementer uavhengig av hverandre er blitt integrert i eksakt samme posisjon i to individer. En insersjon i en gitt posisjon regnes derfor som en unik hendelse som indikerer et felles opphav (identity by descent). Samtidig representerer fraværet av Alu-elementet den opprinnelige tilstanden (ancestral state) for posisjonen man undersøker (14).
Disse egenskapene er utnyttet i ulike fylogenetiske undersøkelser. En studie av Hamdi og medarbeidere (15) illustrerer hvordan Alu-elementer er benyttet for å kartlegge slektskap mellom ulike primater. Ved å undersøke for tilstedeværelse av Alu-elementer i ortologe posisjoner kunne to arter klassifiseres som nærere beslektet enn det slektskapet de hadde til en tredje art fordi de hadde et Alu-element i en posisjon der den tredje arten manglet det. Slektskap ble med andre ord basert på at to arter hadde fått en felles karakteristisk markør, aldri på at de manglet markøren. Den samlede informasjonen fra 15 Alu-elementer støttet kun ett fylogenetisk slektstre for de åtte arter som ble undersøkt, i samsvar med at Alu-insersjoner er unike hendelser.
Alu-elementer kan benyttes på liknende måter for å kartlegge rekkefølgen av genomiske rearrangementer. Ved slike undersøkelser kan man få ytterligere informasjon ved å sammenlikne sekvensdiversitet mellom de enkelte elementene. Veksthormongener (16) og HLA-gener (17) er gode eksempler på historiske rearrangementer i det humane genom som er kartlagt ved hjelp av informasjon fra Alu-elementinsersjoner.
Bialleliske Alu-polymorfismer
Enkelte Alu-elementer som er blitt integrert inn i nye posisjoner de siste 30 000 år, er ikke fiksert i det humane genom, men kan enten være til stede eller ikke være til stede. Slike bialleliske Alu-polymorfismer kan benyttes til å studere populasjonshistorie og genetisk diversitet (18).
Et Alu-element på den ikke-rekombinerende delen av Y-kromosomet (Y-Alu-polymorphic element, YAP) er en biallelisk polymorfisme som er spesielt mye benyttet i kartleggingen av historisk slektskap mellom menn. YAP er høyfrekvent i asiatiske populasjoner, og siden tilstedeværelsen av dette Alu-elementet indikerer historisk slektskap, er det blitt en viktig markør for å kartlegge historiske migrasjoner i ulike folkegrupper (19, 20).
Diskusjon
Den tilsynelatende parasittiske amplifiseringen av Alu-elementer har frembrakt markører i det humane genom som kan benyttes for en rekke ulike formål. Hvorvidt den enorme mengden Alu-elementer i primatgenomer har en eller annen generell funksjon, for eksempel ved stressrespons, er fremdeles et åpent spørsmål (4).
Elementene kan bidra til genetisk sykdom på flere måter. De fleste eksemplene i denne artikkelen illustrerer mekanismer der det er en direkte sammenheng mellom Alu-element og sykdom. I tillegg kan sannsynligvis insersjon av slike elementer nær gener gi disposisjon for sykdom ved å påvirke ekspresjonen av disse genene.
Det er sannsynligvis en underrapportering av Alu-elementer som genererer sykdom. Dette skyldes at de mest benyttede metodene i mutasjonsundersøkelser (SSCP, DGGE, DNA-sekvensering) er designet for å oppdage baseparmutasjoner i eksoner. Tap av et helt ekson pga. en Alu-rekombinasjon vil derfor ofte ikke bli oppdaget (21, 22). Dersom man ønsker å finne alle mutasjoner i et gen, bør derfor metoder som proteintrunkeringstest (testet på mRNA) og kvantitativ PCR-undersøkelse vurderes, slik at også mutasjoner som er forårsaket av Alu-elementer blir oppdaget.
Jeg takker Karen Helene Ørstavik for konstruktiv kritikk av manuskriptet.
Oppgitte interessekonflikter: Ingen
- 1.
Batzer MA, Deininger PL. Alu repeats and human genomic diversity. Nat Rev Genet 2002; 3: 370 – 9.
- 2.
Lander ES, Linton LM, Birren B et al. Initial sequencing and analysis of the human genome. Nature 2001; 409: 860 – 921.
- 3.
Deininger PL, Batzer MA. Alu repeats and human disease. Mol Genet Metab 1999; 67: 183 – 93.
- 4.
Smit AF. Interspersed repeats and other mementos of transposable elements in mammalian genomes. Curr Opin Genet Dev 1999; 9: 657 – 63.
- 5.
PubMed søkemotor for litteraturdatabasen Medline. http://www.ncbi.nlm.nih.gov/entrez/query.fcgi
- 6.
Roeder GS. Meiotic chromosomes: it takes two to tango. Genes Dev 1997; 11: 2600 – 21.
- 7.
Deininger PL, Jolly DL, Rubin CM et al. Base sequence studies of 300 nucleotide renatured repeated human DNA clones. J Mol Biol 1981; 151: 17 – 33.
- 8.
Nystrom-Lahti M, Kristo P, Nicolaides NC et al. Founding mutations and Alu-mediated recombination in hereditary colon cancer. Nat Med 1995; 1: 1203 – 6.
- 9.
Gebow D, Miselis N, Liber HL. Homologous and nonhomologous recombination resulting in deletion: effects of p53 status, microhomology, and repetitive DNA length and orientation. Mol Cell Biol 2000; 20: 4028 – 35.
- 10.
Challem JJ, Taylor EW. Retroviruses, ascorbate, and mutations, in the evolution of Homo sapiens. Free Radic Biol Med 1998; 25: 130 – 2.
- 11.
Nelson DL, Ledbetter SA, Corbo L et al. Alu polymerase chain reaction: a method for rapid isolation of human-specific sequences from complex DNA sources. Proc Natl Acad Sci USA 1989; 86: 6686 – 90.
- 12.
Shih C, Weinberg RA. Isolation of a transforming sequence from a human bladder carcinoma cell line. Cell 1982; 29: 161 – 9.
- 13.
Tyler MG, Kirby LT, Wood S et al. Human blood stain identification and sex determination in dried blood stains using recombinant DNA techniques. Forensic Sci Int 1986; 31: 267 – 72.
- 14.
Shedlock AM, Okada N. SINE insertions: powerful tools for molecular systematics. Bioessays 2000; 22: 148 – 60.
- 15.
Hamdi H, Nishio H, Zielinski R et al. Origin and phylogenetic distribution of Alu DNA repeats: irreversible events in the evolution of primates. J Mol Biol 1999; 289: 861 – 71.
- 16.
Toda Y, Tomita M. Alu elements as an aid in deciphering genome rearrangements. Gene 1997; 205: 173 – 6.
- 17.
Del Pozzo G, Guardiola J. A SINE insertion provides information on the divergence of the HLA-DQA1 and HLA-DQA2 genes. Immunogenetics 1990; 31: 229 – 32.
- 18.
Romualdi C, Balding D, Nasidze IS et al. Patterns of human diversity, within and among continents, inferred from biallelic DNA polymorphisms. Genome Res 2002; 12: 602 – 12.
- 19.
The Y-chromosome consortium. A nomenclature system for the tree of human Y-chromosomal binary haplogroups. Genome Res 2002; 12: 339 – 48.
- 20.
Hammer MF. A recent insertion of an alu element on the Y chromosome is a useful marker for human population studies. Mol Biol Evol 1994; 11: 749 – 61.
- 21.
Wang Y, Friedl W, Sengteller M et al. A modified multiplex PCR assay for detection of large deletions in MSH2 and MLH1. Hum Mutat 2002; 19: 279 – 86.
- 22.
Wang Y, Friedl W, Lamberti C et al. Hereditary nonpolyposis colorectal cancer: frequent occurrence of large genomic deletions in MSH2 and MLH1 genes. Int J Cancer 2003; 103: 636 – 41.